Fortschritte in der biomedizinischen Forschung sind nicht denkbar ohne Fortschritte in der Identifizierung und Analyse der in lebenden Zellen enthaltenen Proteine. Als Schlüsseltechnik hat sich hier die Massenspektrometrie herausgestellt. Sie setzt jedoch voraus, dass die Moleküle einzeln und in elektrisch geladener Form in der Gasphase vorliegen. Bei der dafür notwendigen Präparation können die empfindlichen Makromoleküle leicht zerstört werden. Eine neue, sehr schonende Methode haben nun Forscher am MPQ in Zusammenarbeit mit der Hochschule Esslingen entwickelt. Bereits vor einigen Jahren ließen die MPQ-Forscher ein Verfahren patentieren, bei dem Strahlen aus neutralen Molekülclustern Atome und Moleküle von beliebigen Oberflächen abtragen.
Wie Dr. Christoph Gebhardt, Dr. Anna Tomsic, Dr. Hartmut Schröder, Prof. Michael Dürr und Prof. Karl L. Kompa (emeritierter Direktor am MPQ und Leiter der Gruppe "Laserchemie") in der Zeitschrift Angewandte Chemie (siehe unten) berichten, ist es ihnen nun erstmals gelungen, dieses Verfahren der Desorption mittels transienter Matrix erfolgreich auf große Biomoleküle wie z.B. Insulin anzuwenden. Das Verfahren zeichnet sich dabei u. a. durch die sehr einfache Probenpräparation sowie den sehr schonenden Ablöseprozess aus, bei dem auch große Moleküle vollständig intakt bleiben, aus.
Die Gruppe "Laserchemie" von Emeritus Prof. Karl-Ludwig Kompa arbeitet schon lange mit Clustern aus polaren Molekülen (d.h. die Moleküle haben zwei Ladungsschwerpunkte, sind aber nach außen neutral), die sich bilden, wenn das Ausgangsgas ins Vakuum expandiert. Diese schneeballähnlichen Gebilde bestehen aus 1000 bis 10000 Einzelmolekülen, was einem Durchmesser von etwa 10 Nanometern entspricht. Der Zusammenhalt der Moleküle im Cluster ist allerdings nur schwach. Getrieben durch einen Gasjet aus Helium erreichen sie die zu untersuchende Oberfläche mit etwa dreifacher Schallgeschwindigkeit.
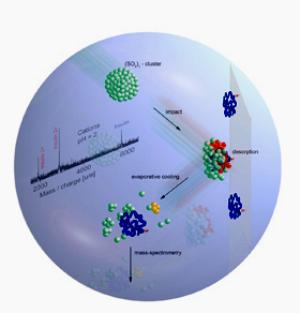
Im vorliegenden Experiment schießen die Forscher solche Cluster aus Schwefeldioxidmolekülen auf Festkörperoberflächen, auf die sie zuvor gelöste Biomoleküle als Film aufgebracht hatten. Aufgrund seines polaren Charakters kann ein Cluster während dieses Stoßes ein Biomolekül, das sich aus dem Lösungsmittel ein positiv geladenes Wasserstoffatom geschnappt hat, mit sich reißen (siehe auch Abbildung). Für einen Zeitraum von nur 10 Pikosekunden - das ist ein Hundertstel von einem Millionstel von einer Sekunde - heizt sich der Cluster im Stoß mit der Oberfläche auf. Danach zerplatzt der heiße Cluster, während das abgelöste Biomolekül - einzeln und elektrisch geladen - in einem herkömmlichen Massenspektrometer analysiert werden kann. "Wir können uns die Cluster als die kleinste denkbare Einheit vorstellen, in denen chemische Reaktionen wie in einem mikroskopischen Reagenzglas ablaufen können. Der Stoß mit der Oberfläche zerstört das Reagenzglas, und wir können die Reaktionsprodukte, sofern sie elektrisch geladen sind, bequem analysieren. Was wir hier machen, ist praktisch Nanochemie", erklärt Dr. Hartmut Schröder.
Auf diese Weise äußerst schonend präpariert, analysierten die Wissenschaftler eine Reihe von Biomolekülen wie Bombesin, Angiotensin oder Insulin mit Hilfe der Flugzeit-Massenspektrometrie. Bei diesem Verfahren wird die Masse aus der Zeit abgeleitet, die das geladene Molekül nach Beschleunigung in einem elektrostatischen Feld zum Durchfliegen einer gegebenen Wegstrecke benötigt. Die Messungen zeigten, dass auf diese Weise nicht nur die einzelnen Biomoleküle identifiziert werden können, sondern vor allem auch, dass die desorbierten Biomoleküle den heftigen Cluster-Oberflächenstoß völlig unbeschadet überstehen. "Dabei zeichnet sich die Methode schon heute durch eine sehr hohe Empfindlichkeit im Femtomolbereich aus", betont Prof. Karl-Ludwig Kompa. "Es gilt nun, diese Methode vom Forschungsstadium in Richtung Anwendung weiterzuentwickeln", ergänzt Prof. Michael Dürr. "Dafür müssen wir sowohl die der Desorption zugrunde liegenden Prozesse besser verstehen, als auch die technischen Voraussetzungen für eine weitere Steigerung der Nachweisempfindlichkeit schaffen." Das hier beschriebene Verfahren soll deshalb im Rahmen der bestehenden Kooperation zwischen dem MPQ und der Hochschule Esslingen weiter erforscht werden.
[OM]
Zusatzinformationen:
Christoph R. Gebhardt, Anna Tomsic, Hartmut Schröder, Michael Dürr, Karl L. Kompa:
Matrix-Free Formation of Gas-Phase Biomolecular Ions by Soft Cluster-Induced Desorption.
In: Angewandte Chemie International Edition; online erschienen am 12. März 2009, DOI 10.1002/anie.200804431
Quelle: Max-Planck-Institut für Quantenoptik, Garching
Aktualisiert am 08.04.2009.
Permalink: https://www.internetchemie.info/news/2009/apr09/transiente-matrix-desorption-zur-massenbestimmung-von-biomolekülen.php
© 1996 - 2026 Internetchemie ChemLin