Wissenschaftler am Max-Planck-Institut für Entwicklungsbiologie in Tübingen haben die Struktur eines Proteins (L1ORF1p) aufgeklärt, das von einem parasitären genetischen Element codiert wird und für dessen Mobilität essentiell ist. Dies ermöglicht neue Erkenntnisse über Verwandtschaftsbeziehungen zwischen Retrotransposons und Retroviren und wahrscheinlich auch über Evolutionsprozesse bei Menschen und Tieren. Außerdem könnten über den Mechanismus des LINE-1-Retrotransposons in Zukunft gezielt genetische Information in das Erbgut eingeschleust werden. Dies wäre eine Alternative zu den weniger zielgerichteten Methoden, die auf einem retroviralen Mechanismus basieren.
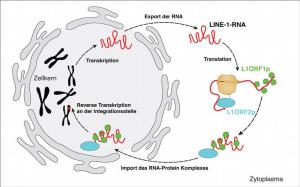
Das so genannte LINE-1-Retrotransposon ist ein mobiles genetisches Element, das sich vermehren und an verschiedenen Stellen in die chromosomale DNA einbauen kann. Dadurch wird der genetische Code an der Integrationsstelle gestört. Dies kann schwerwiegende Konsequenzen für den Organismus haben, führt aber andererseits auch zu genetischer Variation, einer zwingenden Vorraussetzung für die molekulare Evolution der Arten. Die Strukturaufklärung des L1ORF1p-Proteins erlaubt es nun, den Mechanismus der LINE-1-Mobilisierung wesentlich genauer zu untersuchen.
Das LINE-1-Retrotransposon ist ein mobiles Gen, das sich in der Evolutionsgeschichte des menschlichen Genoms massiv vermehrt hat. Gegenwärtig besteht ungefähr 17 Prozent unserer DNA aus LINE-1-Sequenzen. Dies ist ein riesiger Anteil, wenn man berücksichtigt, dass unsere etwa 30.000 Proteine von weniger als fünf Prozent der DNA kodiert werden. Das LINE-1-Retrotransposon kann sich nicht nur selbst vermehren und in die chromosomale DNA integrieren, es ist auch für die Integration von ungefähr einer Million Alu-Sequenzen (ein weiteres parasitäres Gen) verantwortlich. Alu-Sequenzen, die nur bei den höheren Primaten auftreten, machen weitere zehn Prozent unseres Genoms aus. Die Eingliederung von LINE-1 und Alu-Elementen ist ein fortdauernder Prozess und es wird geschätzt, dass mehr als jedes zwanzigste Neugeborene mindestens eine neue Integration eines solchen Elementes aufweist.
Als Konsequenz gibt es kaum ein menschliches Gen, welches nicht schon einmal durch die Integration eines LINE-1 oder Alu-Elementes betroffen wurde. "Es ist kaum vorstellbar, dass die massive Integration von LINE-1 und Alu-Sequenzen ohne Auswirkungen auf die Evolution des Menschen blieb. Daher ist es erstaunlich, wie wenig bisher über den Mechanismus der Retrotransposition und über die dabei involvierten Proteine und Nukleinsären bekannt ist", sagte Oliver Weichenrieder vom Max-Planck-Institut für Entwicklungsbiologie. Die Forscher versuchen nun, die beteiligten Moleküle biochemisch zu charakterisieren und ihre molekularen Strukturen aufzuklären. Auf diese Art schaffen sie die Grundlagen für eine detaillierte Funktionsanalyse und können Ähnlichkeiten zu bereits bekannten Proteinen aufzeigen, insbesondere Ähnlichkeiten, die aufgrund eines simplen Vergleiches der entsprechenden Aminosäuresequenzen nicht offensichtlich sind.
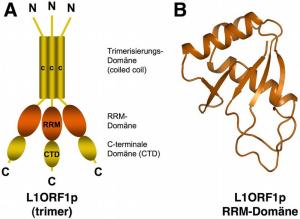
In der veröffentlichten Arbeit [siehe unten] charakterisieren Elena Khazina und Oliver Weichenrieder eines von zwei Proteinen, die vom LINE-1-Retrotransposon kodiert werden. Dieses sogenannte L1ORF1p-Protein bindet an LINE-1-RNA, die von einem LINE-1-Element in der DNA abgeschrieben wurde. L1ORF1p unterstützt dann wohl das anschließende Rückkopieren der LINE-1-RNA in DNA. Dieser Prozess findet direkt an der Stelle statt, wo sich das neue LINE-1 Element ins Genom integriert. Die Wissenschaftler konnten zeigen, dass das L1ORF1p Protein aus drei Teilen besteht. Der erste Teil führt dazu, dass sich immer drei L1ORF1p Proteine zu einem Trimer zusammenlagern. Die anderen beiden Teile sind für die Bindung an die LINE-1-RNA notwendig. "Besonders überraschend war die Identifizierung einer sogenannten RRM-Domäne im mittleren Teil des Proteins, da bisher angenommen wurde, dass dieser Teil des Proteins eher unstrukturiert ist", sagte Elena Khazina. "Unsere Kristallstruktur weist die Existenz dieser Domäne eindeutig nach. Außerdem haben wir RRM-Domänen inzwischen auch in anderen Retrotransposons in verschiedenen Tier- und Pflanzenarten gefunden", so die Strukturbiologin. RRM-Domänen (RNA Recognition Motif; RNA-Erkennungs-Motiv) kommen in der Zelle häufig vor, hauptsächlich in RNA-bindenden Proteinen. Die Existenz einer RRM-Domäne in L1ORF1p erklärt nun warum das L1ORF1p-Protein an die LINE-1-RNA bindet und wie das im Detail passieren könnte.
Das Wissen um die Struktur des L1ORF1p-Proteins bietet einen neuen Blickwinkel und eine gute Grundlage, um in Zukunft diejenigen zellulären Prozesse zu untersuchen, die das LINE-1-Element für seine eigene Vermehrung ausnützt und rekrutiert, beziehungsweise auch diejenigen Mechanismen, welche die Zelle zur Verfügung hat um die übermäßige Vermehrung von Retrotransposons zu unterbinden.
Zusatzinformationen:
Elena Khazina und Oliver Weichenrieder:
Non-LTR retrotransposons encode noncanonical RRM domains in their first open reading frame.
In: Proceedings of the National Academy of Sciences; PNAS, online erschienen am 12. Januar 2009, DOI 10.1073/pnas.0809964106
Quelle: Max-Planck-Institut für Entwicklungsbiologie
Aktualisiert am 20.01.2009.
Permalink: https://www.internetchemie.info/news/2009/jan09/molekulare-parasiten-im-genom.php
© 1996 - 2026 Internetchemie ChemLin