Sie veröffentlichen ihre Erkenntnisse in der Wissenschaftszeitschrift Nature vom 2. Dezember 2010 [siehe Hinweis unten].
Die Biosynthese von Proteinen findet an großen Molekülkomplexen statt, den so genannten Ribosomen. Sie fungieren als Montageplattformen, an denen Ausgangsmaterialien und Werkzeuge zusammengeführt werden. Damit funktionsfähige Eiweißverbindungen entstehen, müssen die Aminosäuren genau so angeordnet werden, wie es genetisch vorgegeben ist.
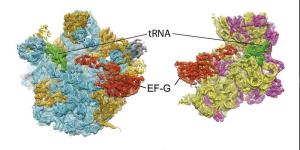
Als Verbindungsstück zwischen der genetischen Vorlage und dem entsprechenden Protein kommt die so genannte tRNA zum Einsatz: Sie trägt jeweils eine bestimmte Aminosäure und verfügt zugleich über eine Bindungssequenz, die exakt zu definierten Stellen der Vorlage passt. Auf diese Weise gelangt jede Aminosäure automatisch an die für sie vorgesehene Position. Sobald ein tRNA-Molekül seine Aminosäure an die wachsende Kette eines Proteins abgegeben hat, wandert es entlang des Ribosoms weiter, um Platz für die nächste tRNA zu machen. Dabei bildet das Ribosom einen Komplex mit einem Enzym namens EF-G, das den Transportprozess erleichtert.
Die beteiligten Wissenschaftler sind nun der Frage nachgegangen, wie der Ortswechsel der tRNA-Moleküle im Detail vor sich geht - ein anspruchsvolles Unterfangen, weil die Bewegung sehr schnell vor sich geht, so dass sie nur schwer zu beobachten ist. Die internationale Arbeitsgruppe rekonstruierte die aufeinander folgenden Zustände des Ribosoms, indem ein Team um Professor Dr. Christian Spahn an der Charité in Berlin die Methode der dreidimensionalen Kryo-Elektronenmikroskopie nutzte. "Bei diesem Verfahren werden die Ribosomen in flüssigem Ethan bei - 192° Celsius schockgefroren und mehrere 100.000 zweidimensionale Einzelbilder in zwei dreidimensionale Rekonstruktionen zurückprojiziert", erläutern die Wissenschaftler.
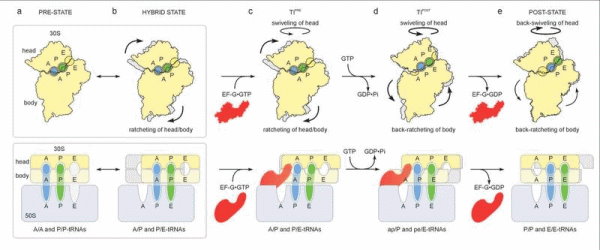
Für die tRNA lassen sich drei Positionen unterscheiden: An der ersten bindet die tRNA ans Ribosom, wobei sie eine Aminosäure trägt (A-Position); an der zweiten hat sie die Aminosäure abgegeben (P); und an der dritten verlässt die tRNA das Ribosom (E). Den Autoren ist es nun gelungen, einen neuartigen Übergangszustand zu identifizieren, bei dem sich die tRNA zwischen zwei dieser Positionen befindet.
Wie die Forscher darüber hinaus feststellten, geht dieser Transport mit festgelegten Bewegungen des Ribosoms einher: Dessen beide Untereinheiten vollführen gegenläufige Drehungen und schieben dadurch die tRNA von einer Position zur nächsten. Hierbei gewährleistet das EF-G-Enzym, dass die Bewegungsrichtung beibehalten wird - wie ein Türstopper, der das Zurückschwingen verhindert.
"Unsere Einblicke in die Struktur der tRNA-Zustände legen ein Modell nahe, bei dem der tRNA-Transport durch Drehung, Einrasten und Entsperren des Ribosoms erleichtert werden", fassen die Forscher ihre Ergebnisse zusammen. "Diese Befunde erlauben unmittelbare strukturelle und mechanistische Einsicht in die Zwischenprodukte, die an der universell konservierten Translokation beteilgt sind."
(Bildquelle: Philipps-Universität Marburg / AG Hartmann)
An der Publikation in "Nature" sind neben Roland Hartmann und seinem Mitarbeiter Andreas Ratje weitere Wissenschaftler aus Berlin, München, Frankfurt und den USA beteiligt. Hartmanns Arbeitsgruppe am Institut für Pharmazeutische Chemie der Philipps-Universität beschäftigt sich vor allem mit der Struktur und Funktion katalytischer und regulatorischer RNA-Moleküle.
Pressemitteilung der Charité-Universitätsmedizin Berlin
Biologische Zahnräder treiben zelluläre Maschine zur Eiweißproduktion an
Forschergruppe löst Grundproblem der Molekularbiologie
Wissenschaftlerinnen und Wissenschaftler der Charité - Universitätsmedizin Berlin haben in Zusammenarbeit mit Arbeitsgruppen an mehreren deutschen und US-amerikanischen Forschungsstandorten eine Schlüsselfrage bei der Herstellung von Eiweißen in der Zelle beantworten können. Eine mögliche Anwendung dieser Entdeckung sehen die Forscher, deren Arbeit im führenden Fachmagazin "Nature"* veröffentlicht wurde, in der Entwicklung von verbesserten Antibiotika.
Eiweiße entstehen im Zusammenspiel zweier Ribonukleinsäuren (RNS) in den Ribosomen, den "Eiweißfabriken" der Zelle. Dabei wird der Bauplan der Eiweiße, die sogenannte Boten-RNS, wie ein Magnetfilm an der Nahtstelle zwischen den beiden Untereinheiten des Ribosoms abgelesen. Anschließend werden die Eiweiße kettenartig aus Aminosäuren aufgebaut. Leser der Boten-RNS und gleichzeitig Träger der Aminosäuren sind die Transfer-RNS. Diese transportieren die zum Aufbau der Eiweiße benötigten Aminosäuren solange zum Ort der Synthese, bis der Bauplan das Ende dieser Arbeit signalisiert. Damit ist der genetische Code, der in der Abfolge der Nukleinsäuren gespeichert ist, von der Nukleinsäurewelt in ein Produkt der Proteinwelt übersetzt worden. Das fertige Eiweiß verlässt dann über einen Tunnel das Ribosom. Unklar war bislang, wie die Boten-RNS und die Transfer-RNS durch das Ribosom bewegt werden.
Den Forschern und Forscherinnen aus Berlin, Marburg, München, Frankfurt/Main, Los Alamos, San Diego, Tallahassee und Houston gelang es nun zu dokumentieren, dass die Bewegung durch gegenläufige Drehungen der beiden Untereinheiten des Ribosoms zustande kommt. "Dabei wird Wärmeenergie genutzt. Diese sorgt für die interne Bewegung des Ribosoms, und dadurch wiederum werden Transfer- und Boten-RNS durch das Ribosom bewegt", erklärt Prof. Christian Spahn, Direktor des Instituts für Medizinische Physik und Biophysik am Campus Charité Mitte, der die multinationale Forschungsgruppe koordinierte. Vergleichen lässt sich die Arbeit der Untereinheiten des Ribosoms mit einer mechanischen Ratsche. Eine Schlüsselstellung nimmt dabei die Kopfdomäne der kleinen Untereinheit ein, welche die Transfer-RNS wie auf einem Fließband herbeitransportiert. Moderiert wird der Vorgang von einem Helferprotein, das so zu sagen als dynamische Sperrklinke dient und die Bewegung in Zielrichtung gewährleistet.
Die Lösung dieses langjährigen Problems der Molekularbiologie wurde durch die strukturelle Untersuchung mittels dreidimensionaler Kryo-Elektronenmikroskopie ermöglicht. Bei dieser Methode werden die Ribosomen in flüssigem Ethan bei minus 192 Grad Celsius schockgefroren und mehrere 100.000 zweidimensionale Einzelbilder von Ribosomen nach Sortierung in zwei dreidimensionale Rekonstruktionen zurückprojiziert.
Zusatzinformationen:
Andreas H. Ratje, Justus Loerke, Aleksandra Mikolajka, Matthias Brünner, Peter W. Hildebrand, Agata L. Starosta, Alexandra Dönhöfer, Sean R. Connell, Paola Fucini, Thorsten Mielke, Paul C. Whitford, José N. Onuchic, Yanan Yu, Karissa Y. Sanbonmatsu, Roland K. Hartmann, Pawel A. Penczek, Daniel N. Wilson und Christian M. T. Spahn:
Head swivel on the ribosome facilitates translocation by means of intra-subunit tRNA hybrid sites.
In: Nature; 468, 713 - 716, online veröffentlicht am 01. Dezember 2010, DOI 10.1038/nature09547
Quelle: Philipps-Universität, Marburg
Aktualisiert am 08.12.2010.
Permalink: https://www.internetchemie.info/news/2010/dec10/zellulaere-maschinerie-der-proteinproduktion.php
© 1996 - 2026 Internetchemie ChemLin