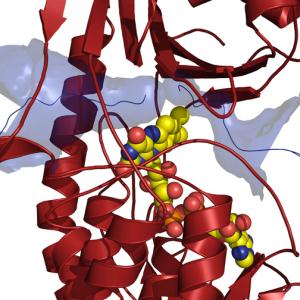
Forscher der Universität Greifswald haben jetzt die Struktur des Proteins Monooxygenase TetX aufgeklärt, das Bakterien eine Resistenz gegen Tetracyclin-Antibiotika vermittelt. Damit wurde erstmals ein Schlüssel zum Verständnis eines Resistenzmechanismus gefunden, noch bevor die klinische Anwendung eines Antibiotikums wirkungslos wird.
Ein entsprechender Artikel wurde in der wissenschaftlichen Fachzeitschrift FEBS Letters veröffentlicht [siehe unten].
In enger Kooperation mit der kanadischen Gruppe um Prof. Gerard D. Wright (Institute for Infectious Disease Research, McMaster University, Hamilton, Canada) und Dr. Manfred S. Weiss (Helmholtz-Zentrum Berlin für Materialien und Energie) hat die Greifswalder Arbeitsgruppe von Prof. Winfried Hinrichs im Institut für Biochemie mit Methoden der Proteinkristallographie die dreidimensionale Struktur des Resistenzproteins aufgeklärt.
Die Entwicklung und Anwendung von Antibiotika zur Behandlung von bakteriellen Infektionskrankheiten wird seit langer Zeit durch die Ausbreitung klinisch problematischer Resistenzmechanismen immer schwieriger und kostspieliger. Wird ein neuer Wirkstoff nach jahrelanger Entwicklung auf den Markt gebracht, dauert es nicht lange, bis resistente Mikroorganismen bekannt werden - ein ewiges Wettrüsten.
Kürzlich wurde die Tetracyclin-Variante (Tigecyclin) in die klinische Anwendung aufgenommen. Von diesem Antibiotikum ist bekannt, dass es gegen resistente Bakterien hochwirksam ist. Leider ist auch hier bereits ein bakterieller Resistenzmechanismus bekannt, der momentan noch keine Rolle bei der Anwendung in der Human- und Tiermedizin spielt. Aber es steht zu befürchten, dass sich dieser Mechanismus auf problematische Krankheitskeime ausbreiten wird. Diese Resistenz beruht auf einem Enzym, der Monooxygenase TetX. Es versetzt die Bakterien in die Lage, alle medizinisch relevanten Tetracycline gezielt abzubauen.
Die röntgen-kristallographischen Untersuchungen wurden an Synchrotron-Strahlungsquellen in Hamburg (EMBL c/o DESY) und Berlin (BESSY) durchgeführt. Diese Arbeiten sind Teil der Doktorarbeit von Dipl.-Biochem. Gesa Volkers aus Greifswald. Ihre kürzlich publizierten Strukturanalysen geben Einblick in die atomare Struktur des Enzym-Moleküls und zeigen, wie Tetracycline erkannt werden.
Damit ist der einzigartige Fall eingetreten, dass ein Schlüsselschritt eines Resistenzmechanismus auf molekularer Ebene bekannt ist, noch bevor das Antibiotikum wirkungslos wird. Auf dieser Basis könnte es möglich werden, Konzepte zu entwickeln, wie neue Tetracyclin-Moleküle umzubauen sind, damit sie gegen TetX stabil sind und wieder antibiotische Wirkung haben ("drug design").
Mit diesem Projekt konnte die Arbeitsgruppe von Prof. Hinrichs bereits zum zweiten Mal strukturbiologische Grundlagen zu Resistenzmechanismen gegen Tetracycline liefern.
Zusatzinformationen:
Gesa Volkers, Gottfried J. Palm, Manfred S. Weiss, Gerard D. Wright, Winfried Hinrichs:
Structural basis for a new tetracycline resistance mechanism relying on the TetX monooxygenase.
In: FEBS Letters; Volume 585, Issue 7, Pages 1061 - 1066, 06. April 2011, DOI 10.1016/j.febslet.2011.03.012
Quelle: Ernst-Moritz-Arndt-Universität, Greifswald
Aktualisiert am 21.04.2011.
Permalink: https://www.internetchemie.info/news/2011/apr11/monooxygenase-tetx.php
© 1996 - 2026 Internetchemie ChemLin