Mit Hilfe der Vibrationsspektroskopie und biomolekularer Simulationen haben die Bochumer Forscher somit erstmals vollständig den Mechanismus aufgeklärt, mit dem ein Membranprotein Protonen durch die Zellmembran pumpt. Sie zeigten, dass proteingebundene Wassermoleküle hierfür eine entscheidende Rolle spielen. Ihre Ergebnisse wurden für die early Edition von PNAS ausgewählt [siehe unten].
Proteingebundene Wasser sind entscheidend
Bestimmte Proteine können Protonen von einer Seite der Zellmembran (Aufnahmeseite) auf die andere transportieren (Abgabeseite), was einen zentralen Prozess der Energieumwandlung in der Biologie darstellt. In früheren Veröffentlichungen in Nature und Angewandte Chemie konnten die Forscher des Lehrstuhls für Biophysik bereits zeigen, dass die proteingebundenen Wassermoleküle auf der Abgabeseite im Ruhezustand optimal angeordnet sind, um Protonen abzugeben.
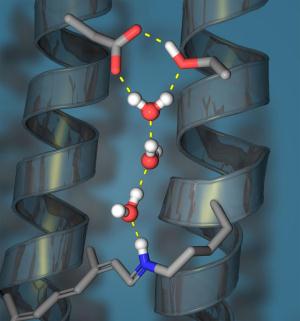
"Die Protonen werden wie bei umfallenden Dominosteinen vom Protein angestoßen und infolgedessen heraus befördert", erklärt Gerwert. Unklar blieb jedoch, wie das Protein wieder in den Ausgangszustand zurückgesetzt wird, um einen neuen Pumpzyklus starten zu können. Um die abgegebenen Protonen zu ersetzen, müssen an der anderen Seite des Proteins neue Protonen aufgenommen werden. Die Bochumer Forscher fanden heraus, dass sich zu diesem Zweck an der Aufnahmeseite eine Kette von gerade mal drei Wassermolekülen für nur wenige Tausendstel einer Sekunde bildet, um die Protonen ins Proteininnere zu leiten.
Das Wasser gibt die Richtung vor
Das Protein schlägt dabei zwei Fliegen mit einer Klappe: In der Abgabephase sind die Wassermoleküle ungeordnet, was einen Protonentransport in die falsche Richtung verhindert. Nur in der Aufnahmephase sind sie korrekt ausgerichtet und können Protonen leiten. Diese Ergebnisse lösen das Rätsel, warum die Protonenleitung an der Aufnahmeseite nur in eine Richtung funktioniert und das Protein somit effektiv und gerichtet pumpen kann. "Die vorliegende Arbeit bildet mit den beiden vorherigen eine Trilogie, die den Protonenpumpzyklus vollständig mit atomarer Auflösung erklärt", resümiert Gerwert.
Experimentelle Physik und theoretische Chemie kombiniert
Um die Prozesse auf Nanoebene mit hoher räumlicher und zeitlicher Auflösung verfolgen zu können, kombinierten die Forscher experimentelle Physik mit theoretischer Chemie. Steffen Wolf simulierte zunächst die strukturellen Änderungen im Protein mittels biomolekularer Computersimulationen (Molekulardynamik-Simulationen). Erik Freier wies die Effekte anschließend experimentell mit einer von Prof. Gerwert entwickelten Form der Vibrationsspektroskopie (zeitaufgelöste step-scan FTIR-Spektroskopie) nach. "Dieses interdisziplinäre Wechselspiel war der Schlüssel zum Erfolg", so Gerwert. "Es hat sich gezeigt, dass die einzelnen Komponenten des Proteins so präzise aufeinander abgestimmt sind wie Zahnräder einer Maschine."
Wie in Wasser so im Protein
Die drei Wassermoleküle werden vom Protein geschickt so angeordnet, dass sie Protonen nach dem aus der physikalischen Chemie bekannten Grotthus-Mechanismus leiten. Diesen Mechanismus beschrieb Nobelpreisträger Manfred Eigen in den fünfziger Jahren, um den sehr schnellen, ungerichteten Protonentransport in Wasser zu erklären. Aus den Bochumer Publikationen ergibt sich nun überraschenderweise, dass Aminosäuren gemeinsam mit proteingebundenen Wassermolekülen diesem sehr schnellen Transport eine Richtung geben können. Gerwerts Team konnte somit die Ergebnisse von Manfred Eigen erweitern und auf die Proteinforschung übertragen.
Lichtenergie effektiv in chemische Energie umwandeln
Die Bochumer Forscher arbeiteten vor allem mit dem Membranprotein Bakteriorhodopsin, mit dem bestimmte Bakterien eine urtümliche Form der Photosynthese ausführen. Bakteriorhodopsin baut ein Protonenkonzentrationsgefälle auf, indem es Protonen aus dem Zellinneren nach außen transportiert. Dieses Gefälle nutzen andere Proteine zur Produktion von ATP, dem universellen Kraftstoff der Zellen. Um die Lichtenergie effektiv zu nutzen, ist es wichtig, dass der Protonentransport eine spezifische Richtung besitzt und dass ein spontaner Rückfluss von Protonen verhindert wird.
Zusatzinformationen:
Erik Freier, Steffen Wolf and Klaus Gerwert:
Proton transfer via a transient linear water-molecule chain in a membrane protein.
In: Proceedings of the National Academy of Sciences; PNAS, online veröffentlicht am 27. Juni 2011, DOI 10.1073/pnas.1104735108
Dr. Steffen Wolf, Erik Freier, Dr. Meike Potschies, Prof. Dr. Eckhard Hofmann, Prof. Dr. Klaus Gerwert:
Gerichteter Protonentransfer in Membranproteinen mittels protonierter proteingebundener Wassermoleküle: eine Protonendiode.
In: Angewandte Chemie; Volume 122, Issue 38, 7041 - 7046, 10. September 2010, DOI 10.1002/ange.201001243
Florian Garczarek, Klaus Gerwert:
Functional waters in intraprotein proton transfer monitored by FTIR difference spectroscopy.
In: Nature; 439, 109-112, 5. Januar 2006, DOI 10.1038/nature04231
Quelle: Ruhr-Universität Bochum, RUB
Aktualisiert am 30.06.2011.
Permalink: https://www.internetchemie.info/news/2011/jun11/proteingebundene-wassermolekuele.php
© 1996 - 2026 Internetchemie ChemLin