Die Photosynthese ist einer der wichtigsten biologischen Prozesse. In Pflanzen ist sie aber weniger effizient als sie sein könnte.
Rotalgen dagegen nutzen einen leicht veränderten Mechanismus und sind daher produktiver.
Forscher vom Max-Planck-Institut für Biochemie (MPIB) haben jetzt ein bisher unbekanntes Helferprotein der Photosynthese in Rotalgen entdeckt. "Wir konnten seine Struktur und seinen faszinierenden Mechanismus entschlüsseln", erläutert Manajit Hayer-Hartl, Gruppenleiterin am MPIB. "Der Vergleich seines Mechanismus mit dem in grünen Pflanzen könnte helfen, effizientere Pflanzen zu entwickeln."
Die Arbeit wurde in Nature und Nature Structural and Molecular Biology veröffentlicht [siehe unten].
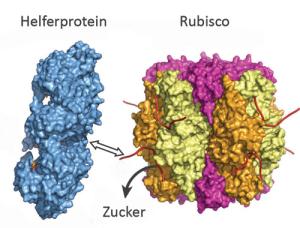
Pflanzen, Algen und Plankton wandeln Kohlenstoffdioxid (CO2) und Wasser in Sauerstoff und Zucker um. Ohne diesen Prozess (Photosynthese) wäre das heutige Leben undenkbar. Somit ist das Schlüsselprotein der Photosynthese, das Forscher kurz Rubisco nennen, eines der wichtigsten Proteine überhaupt. Es bindet CO2 und leitet die Umwandlung in Zucker und Sauerstoff ein.
"Obwohl es so wichtig ist, steckt Rubisco voller Fehler", sagt Manajit Hayer-Hartl, Leiterin der Forschungsgruppe "Chaperonin-vermittelte Proteinfaltung" am MPIB. Ein Problem ist, dass Rubisco falsche Zuckermoleküle bindet, die dann seine Aktivität hemmen. Damit Rubisco seine Arbeit wieder aufnehmen kann, müssen die Zuckermoleküle von einem speziellen Helferprotein (Rubisco Aktivase) entfernt werden. Die Max-Planck-Wissenschaftler haben jetzt entdeckt, dass sich im Laufe der Evolution zwei verschiedene Rubisco Aktivasen in Pflanzen und Rotalgen entwickelt haben. Sie unterscheiden sich in ihrem dreidimensionalen Aufbau und in ihren Arbeitsmechanismen.
Zwei Wege führen zur Wiederherstellung der Rubisco-Aktivität
Die neu entdeckte Rubisco Aktivase in Rotalgen repariert blockierte Rubisco-Proteine, in dem sie an einem Ende des Proteins zieht - wie jemand, der an einem Schnürsenkel zieht. Dadurch öffnet das Helferprotein das aktive Zentrum von Rubisco und setzt so den Zucker frei. Die entsprechende Rubisco Aktivase in Grünpflanzen funktioniert dagegen eher wie ein Eieröffner: Sie quetscht das inaktive Rubisco-Protein und zwingt es so, die Zuckermoleküle frei zu lassen. "Das Verständnis der Struktur und Funktion der beiden Helferproteine könnte helfen Pflanzen und Mikroorganismen herzustellen, die effektiver arbeiten und somit mehr CO2 in Biomasse umwandeln", hofft Manajit Hayer-Hartl.
Zusatzinformationen:
Mathias Stotz, Oliver Mueller-Cajar, Susanne Ciniawsky, Petra Wendler, F Ulrich Hartl, Andreas Bracher und Manajit Hayer-Hartl:
Structure of green-type Rubisco activase from tobacco.
In: Nature Structural and Molecular Biology; online veröffentlicht am 06. November 2011, DOI 10.1038/nsmb.2171
Oliver Mueller-Cajar, Mathias Stotz, Petra Wendler, F. Ulrich Hartl, Andreas Bracher und Manajit Hayer-Hartl:
Structure and function of the AAA+ protein CbbX, a red-type Rubisco activase.
In: Nature; online veröffentlicht am 02. November 2011, DOI 10.1038/nature10568
Andreas Bracher, Amanda Starling-Windhof, F. Ulrich Hartl und Manajit Hayer-Hartl:
Crystal structure of a chaperone-bound assembly intermediate of form I Rubisco.
In: Nature Structural and Molecular Biology; online veröffetnlicht am 17. Juli 2011, DOI 10.1038/nsmb.2090
Quelle: Max-Planck-Institut für Biochemie, Martinsried
Aktualisiert am 08.11.2011.
Permalink: https://www.internetchemie.info/news/2011/nov11/rubisco-aktivase.php
© 1996 - 2026 Internetchemie ChemLin