Ein Verbund von Chemikern der Universität Konstanz entwickelte eine innovative Methode, um Bindungsmechanismen zwischen multivalenten Kohlenhydraten und Proteinen mechanistisch zu untersuchen. Über Abstandsmessungen im Nanometerbereich gewinnen die Forscher direkte Informationen über das Bindungsverhalten der Kohlenhydrate in Lösungen. Bisherige Methoden konnten den Bindungsmodus in Lösung nur indirekt über Berechnungen und Simulationen ableiten, nicht aber strukturelle Daten auf direkte Weise bestimmen.
Die Forschung findet in enger Kopplung an die Konstanzer Graduiertenschule Chemische Biologie statt.
Die Forschungsergebnisse, die unter anderem für die Krebsforschung relevant werden könnten, wurden als Titelbeitrag des Wissenschaftsjournals "Angewandte Chemie" veröffentlicht [siehe unten].
"Kohlenhydrate sind gemeinhin als Nahrungsmittel oder als Gerüst- und Stützsubstanzen bekannt", erklärt Prof. Dr. Valentin Wittmann den Hintergrund des Forschungsprojekts, das in Zusammenarbeit mit der Arbeitsgruppe von Dr. Malte Drescher stattfand. "Es gibt jedoch noch eine ganze Reihe an weiteren Funktionen von Kohlenhydraten: In Form von Zuckerstrukturen sind sie auch auf Zelloberflächen zu finden. Hier wirken sie als Erkennungsstrukturen und dienen zur Signalübermittlung: Über diese Zuckerstrukturen können Zellen miteinander kommunizieren", führt Wittmann weiter aus.
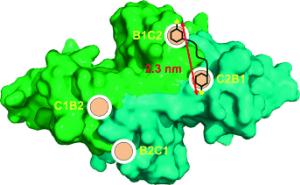
Diese Informationsübermittlung findet über die Anbindung von Kohlenhydrat-Liganden an Proteine statt. Der Prozess erfolgt jedoch nicht ausschließlich über eine einzelne Bindungsstelle, denn häufig dockt der Ligand in brückenförmiger Streckung an gleich mehreren Stellen (multivalent) an das Protein an: Die Bindung wird dadurch fester und für die Informationsübermittlung spezifischer. In der Forschung werden diese Prozesse allerdings meist in kristallinen Proben mithilfe der Röntgenbeugung untersucht. Aufschluss über die tatsächlich vorliegende Bindungsform in Lösung zu gewinnen, war bislang eine Herausforderung.
Die Forschergruppen von Wittmann und Drescher fanden nun eine innovative Methode auf Grundlage der Elektronenspinresonanz-Spektroskopie, um den exakten Bindungsmodus zu bestimmen. An den beiden bindungsfähigen Enden eines sogenannten zweizähnigen Liganden werden Spin-Sonden angebracht. Spektroskopisch wird der Abstand zwischen diesen Sonden gemessen. "Wenn die Ligandenmoleküle sich frei in der Lösung bewegen, dann sind sie flexibel und wir finden eine Mischung langer und kurzer Abstände zwischen den Spin-Sonden", erläutert Malte Drescher. "Bindet der Ligand hingegen in einer brückenartig gestreckten Bindung doppelt an ein Protein an, dann ist plötzlich seine Flexibilität reduziert und wir erhalten in unseren Messwerten keine breite Abstandsverteilung mehr, sondern eine relativ schmale. Über diese Abstandsinformation gewinnen wir eine direkte Strukturinformation über den Liganden und können bestimmen, wie er an das Protein bindet", verrät Drescher das Schlüsselprinzip.
Die gewonnene Methode baut auf Forschungsergebnissen auf, die in Kooperation mit den Arbeitsgruppen der Konstanzer Professoren Dr. Kay Diederichs und Dr. Wolfram Welte (Fachbereich Biologie) sowie Dr. Heiko M. Möller (Fachbereich Chemie) erarbeitet wurden. Die Wissenschaftler wollen ihre Methode nun in weiteren Schritten für die Erforschung von zellulären Wechselwirkungen einsetzen: "Unsere Vorstellung ist, dass wir künftig auf Basis unserer Methode biologisch relevante Systeme untersuchen werden, die zum Beispiel bei der Krebsentstehung eine Rolle spielen", eröffnet Valentin Wittmann einen Ausblick auf die Fortführung der Forschungsarbeiten.
Zusatzinformationen:
Dipl.-Chem. Patrick Braun, M. Sc. Bettina Nägele, Prof. Dr. Valentin Wittmann, Dr. Malte Drescher:
Mechanistische Untersuchung multivalenter Kohlenhydrat-Protein-Wechselwirkungen durch EPR-Spektroskopie.
In: Angewandte Chemie; Volume 123, Issue 36, Seiten 8579 - 8582, 29. August 2011, DOI 10.1002/ange.201101074
Quelle: Universität Konstanz
Aktualisiert am 05.10.2011.
Permalink: https://www.internetchemie.info/news/2011/oct11/multivalente-kohlenhydrat-proteine.php
© 1996 - 2026 Internetchemie ChemLin