Neue Wirkstoffe gegen Krebs oder Malaria aufzuspüren, könnte schon bald einfacher werden - dank eines Computerprogramms, mit dem Forscher vom Max-Planck-Institut für molekulare Physiologie in Dortmund die Suche nach geeigneten Substanzen erleichtern: Das Programm namens Scaffold Hunter - scaffold bedeutet im Englischen Grundgerüst und hunter ist der Jäger - dient als Navigationshilfe durch den chemischen Strukturraum. Es erzeugt Karten chemisch verwandter Strukturen und verknüpft sie mit der biologischen Aktivität, das heißt ihrem Potenzial, an Proteine zu binden - vor allem an solche, die medizinisch relevant sind. Mit Hilfe der neuen Methode haben die Max-Planck-Wissenschaftler zusammen mit Kollegen von den Universitäten Frankfurt und Eindhofen sowie der University of New Mexiko Substanzen identifiziert, die als Kandidaten in Frage kommen könnten, um daraus Wirkstoffe für die Krebstherapie und die Behandlung von Malaria zu entwickeln (siehe unten).
Die Dimensionen des chemischen Strukturraums sind unvorstellbar: Mit der Gesamtzahl aller denkbaren chemischen Strukturen enthält er schätzungsweise bis zu 10160 unterschiedliche Moleküle. Das ist eine Zahl, die zwei Zeilen einer eng beschriebenen Schreibmaschinenseite füllt. Doch nur ein Teil davon - laut Schätzungen 1060 Moleküle - eignet sich möglicherweise als Wirkstoffe. Diese Inseln biologischer Aktivität im Ozean aller potenziellen Verbindungen ausfindig zu machen, gestaltet sich schwierig.
"Organische Synthese kann den chemischen Strukturraum nicht vollständig ermessen" erklärt Stefan Wetzel, Forscher in der Arbeitsgruppe um Herbert Waldmann am Max-Planck-Institut für molekulare Physiologie in Dortmund. Chemiker können also nicht alle möglichen Verbindungen kochen, um sie zu testen. Um sich in diesem Meer von Möglichkeiten dennoch zu orientieren, stellen die Forscher nun ein Navigationssystem vor. Der Scaffold Hunter erzeugt nach strukturellen Kriterien eine Karte des chemischen Strukturraums und nutzt sie, um biologisch aktive Verbindungen, z.B. Naturstoffe, aufzufinden. Das Programm eignet sich auch, um neue Wirkstoffkandidaten vorauszusagen, die in der Natur nicht vorkommen.
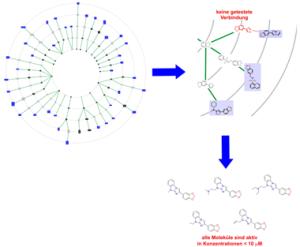
Die Wissenschaftler betrachten jenen medizinisch besonders relevanten Ausschnitt des chemischen Strukturraums, in dem die Moleküle ringförmige Strukturen enthalten. Dabei reduzieren sie die Moleküle jeweils auf ihr charakteristisches Grundgerüst. Diese Strukturen ordnet der Scaffold Hunter nach ihrer Ähnlichkeit in einer Art Stammbaum: Das Programm weist jedem Gerüst übergeordnete kleinere Gerüste zu, indem es nach und nach Ringe entfernt. Das ergibt unzählige Elter-Kind-Beziehungen - strukturell verwandte Moleküle unterschiedlicher Komplexität. Der Clou liegt nun darin, dass chemisch ähnliche Verbindungen sehr wahrscheinlich auch ähnliche biologische Aktivität zeigen.
"Diese strukturbasierten Abstammungslinien bilden die Äste des Baumes", erklärt Stefan Wetzel: "Mit Hilfe des Scaffold Hunters bewegen wir uns nun entlang der Äste, von komplexen hin zu immer einfacheren Strukturen mit möglicherweise ähnlicher Wirkung". So finden die Forscher strukturell einfache Grundgerüste, die als Startpunkte bei der Suche nach neuen Wirkstoffen vielversprechend sind: Die Gerüste können Chemiker dann mit verschiedenen Anhängseln variieren, um den optimalen Wirkstoff zu synthetisieren. Mit dem Scaffold Hunter lassen sich auch bioaktive Moleküle vorhersagen, die in der Natur nicht vorkommen, aber mit hoher Wahrscheinlichkeit ähnliche Aktivität zeigen wie benachbarte natürliche Moleküle. Denn das Programm kreiert und visualisiert auch virtuelle Gerüste. Wie effizient die Methode funktioniert, haben die Forscher gleich bewiesen, und zwar mit der Entdeckung neuer Hemmstoffe der Pyruvatkinase. Die Hemmung dieses Enzyms gilt als vielversprechend bei der Behandlung von Krebs und Malaria.
Noch präziser wird die Suche, wenn die Wissenschaftler bei der Navigation von vornherein Angaben über die biologische Aktivität einfließen lassen - sofern solche Daten vorhanden sind.
In diesem Fall verknüpft der Scaffold Hunter nur solche Gerüste zu Zweigen, die bekanntermaßen gleiche biologische Aktivität zeigen. Diese Äste tragen mit hoher Wahrscheinlichkeit Früchte: Auch in den Lücken zwischen den Substanzen, deren biologische Aktivität bereits bekannt ist, sitzen vermutlich aktive Stoffe. "Wir haben auf diese Weise neue Hemmstoffe für 2-Lipoxygenase und den Östrogenrezeptor alpha ausfindig gemacht", sagt Steffen Renner, früher ebenfalls Forscher am Max-Planck-Institut und jetzt bei der Pharmafirma Novartis tätig. 5-Lipoxygenase ist ein Zielprotein bei der Behandlung von Entzündungen und Blasenkrebs, während der Östrogenrezeptor alpha einen wichtigen Ansatzpunkt bei der Brustkrebstherapie darstellt.
"Der Scaffold Hunter ist eine Schlüsseltechnik mit unzähligen Anwendungsmöglichkeiten" sagt Stefan Wetzel. "Dabei ist das Programm bewusst sehr bedienerfreundlich gehalten, so dass auch Nicht-Experten damit selbständig ihre Daten analysieren können", fügt er hinzu. Die Forscher stellen den Scaffold Hunter im Internet kostenlos zur Verfügung. Auch der Quellcode ist erhältlich - so könnten fortgeschrittene Nutzer das Programm an ihre Bedürfnisse anpassen, um damit noch zielgerichteter durch den chemischen Strukturraum zu navigieren. [EM]
Zusatzinformationen:
Stefan Wetzel, Karsten Klein, Steffen Renner, Daniel Rauh, Tudor I Oprea, Petra Mutzel, Herbert Waldmann:
Interactive exploration of chemical space with Scaffold Hunter.
In: Nature Chemical Biology; online veröffentlicht am 28. Juni 2009, DOI 10.1038/nchembio.187
Steffen Renner, Willem A L van Otterlo, Marta Dominguez Seoane, Sabine Möcklinghoff, Bettina Hofmann, Stefan Wetzel, Ansgar Schuffenhauer, Peter Ertl, Tudor I Oprea, Dieter Steinhilber, Luc Brunsveld, Daniel Rauh, Herbert Waldmann:
Bioactivity-guided mapping and navigation of chemical space.
In: Nature Chemical Biology; online erschienen am 28. Juni 2009, DOI 10.1038/nchembio.188
Quelle: Max-Planck-Institut für molekulare Physiologie, Dortmund
Aktualisiert am 13.08.2009.
Permalink: https://www.internetchemie.info/news/2009/aug09/ozean-der-molekuele.php
© 1996 - 2026 Internetchemie ChemLin